Prof. Dr. med. Christian Jackisch, Prof. Dr. med. Peter J. Wild
Ein Abkürzungsverzeichnis finden Sie am Ende dieses Artikels.
Zusammenfassung
Die Therapie des Mammakarzinoms hat sich in den beiden vergangenen Dekaden von der „one size fits all“ Therapie zu einer individualisierten Therapie weiterentwickelt. Die Analyse von genetischen Veränderungen kann eine therapeutische Relevanz in jeder Krankheitsphase für die betroffene Patientin haben und bietet darüber hinaus auch eine Möglichkeit zur Prävention für nicht betroffene Familienangehörige.
Die Anpassung der Radikalität der lokalen operativen und strahlentherapeutischen Maßnahmen hat zu einer relevanten Morbiditätsreduktion mit Verbesserung der Lebensqualität geführt. Als Grundlage für die systemische adjuvante Therapie ist die Bestimmung des Rückfallrisikos heute mit einer Vielzahl molekularer Marker möglich, die zur risikoadaptierten Auswahl der geeigneten Behandlungen führen. Durch eine erweiterte Diagnostik unter Verwendung von größeren Genpanels können neben der Bestimmung prädiktiver obligatorischer Biomarker auch Informationen über mögliche Resistenzmechanismen sowie über die potenzielle Wirksamkeit von derzeit in klinischen Studien untersuchten Therapieoptionen gewonnen werden. In welchem Umfang molekulare Analysen im individuellen Fall sinnvoll sind, sollte von der lokalen Tumorkonferenz entlang definierter Algorithmen festgelegt werden und durch Angebote der molekularen Tumorboards ergänzt werden, um zukünftig das Monitoring des Krankheitsverlaufs minimal invasiv mit Hilfe der „Liquid Biopsy“ Methode überwachen zu können.
In diesem Beitrag sollen die Grundlagen für die molekulare Diagnostik und deren klinische Konsequenzen für das frühe und metastasierte Mammakarzinom aufgezeigt und diskutiert werden.
Fortbildungsziele
- Welche Marker sind für die adjuvante Therapieauswahl des frühen Mammakarzinoms (early breast cancer, EBC) relevant?
- Welche Biomarker haben eine therapeutische Konsequenz in der Therapie des metastasierten Mammakarzinoms (metastatic breast cancer, MBC)? Wie sind molekulare Analysen in der adjuvanten und metastasierten Erkrankungsphase zu interpretieren?
- Welche Anforderungen werden an die Pathologie und Molekularpathologie gestellt?
- Was ist der Nutzen der klassischen Prognosefaktoren und wie sind diese vor dem Hintergrund der neuen molekularen Analysen und dem Einsatz von Methoden künstlicher Intelligenz (KI) zu interpretieren?
Einleitung
Moderne Sequenzierungstechnologien erweitern die Diagnostik von Krebserkrankungen und ermöglichen einen tieferen Einblick in die Tumorbiologie. Beim Mammakarzinom sind inzwischen mehrere Genveränderungen bekannt, die für die Abklärung des familiären Risikos und der Prognose der betroffenen Patientin etabliert sind. Die Bestimmung prädiktiver Marker mittels molekulargenetischer Methoden sind z. B. beim metastasierten Mammakarzinom für Alterationen der Gene BRCA1/2 (Keimbahn), PIK3CA und optional NTRK1-3 bereits obligat.
Durch eine erweiterte Diagnostik mit größeren Genpanels können der Status prädiktiver obligatorischer Marker sowie Informationen über mögliche Resistenzmechanismen gewonnen werden. Die zunehmende Herausforderung der klinischen Interpretation molekularer Analysen erfordert die interdisziplinäre Zusammenarbeit von Klinikern, Humangenetikern, Pathologen und Molekularpathologen, Psychologen und Software-Experten bis hin zur Nutzung von KI-Algorithmen in molekularen Tumorboards. Dabei werden auch spezialisierte Softwarepakete verwendet, die Molekularpathologen bei der Analyse der genetischen Veränderungen und behandelnde Ärzte bei Therapieentscheidungen unterstützen. Das Mammakarzinom befindet sich damit bereits im Zeitalter der molekular stratifizierten Therapieplanung.
Molekulare Diagnostik beim frühen Mammakarzinom (early breast cancer, EBC)
Die aktuellen Therapieempfehlungen basieren derzeit auf der im Jahr 2000 publizierten Subtypisierung des Mammakarzinoms von Charles Peru [1]. Bei luminalen Karzinomen (HR+/HER2-) hat die Kenntnis des individuellen Rückfallrisikos einen Einfluss auf die Auswahl der adjuvanten Therapie (endokrine Therapie versus chemo-endokrine Therapie). Hier ist die einfache Immunhistochemie nicht mehr ausreichend. Die hier zugelassenen Multi-Gen Assays können beim nodal negativen, frühen Mammakarzinom (HR+/HER2-) den Nutzen einer adjuvanten Chemotherapie prognostisch und zum Teil prädiktiv abschätzen. Sie erfassen die Expression von relevanten Genen und ermöglichen die quantitative Abschätzung des Rückfallrisikos bzw. den wahrscheinlichen Nutzen einer adjuvanten chemo-endokrinen Therapie gegenüber einer alleinigen endokrinen Therapie [2].
Die Organkommission Mamma der Arbeitsgemeinschaft für gynäkologische Onkologie empfiehlt die Nutzung von Multigen-Tests zur individuellen Abschätzung des Rückfallrisikos [3], vgl. im Internet www.ago-online.de → Leitlinien/Empfehlungen → Prognostische und prädiktive Faktoren; Kurzlink: https://tinyurl.com/mr2s8644/.
Ob der Einsatz eines Multigen-Tests jedoch für eine Patientin sinnvoll ist, sollte im Einzelfall nach Abklärung der klinisch-pathologisch relevanten Parameter, die Aufschluss über die Prognose und das individuelle Rezidivrisiko geben können, im Rahmen einer interdisziplinären Tumorkonferenz empfohlen werden. Die prospektiv randomisierten Studienergebnisse der MINDACT-Studie (NCT00433589) und der TAILORx-Studie (NCT00310180) haben dazu geführt, dass beim nodal negativen EBC die Kosten der Tests von den gesetzlichen Krankenkassen übernommen werden. Durch diese Vorgehensweise konnten in jüngerer Zeit ca. 30–40 % von nicht indizierten adjuvanten Chemotherapien eingespart werden.
Bisher wurde die Bestimmung weiterer molekularer Biomarker beim frühen Mammakarzinom nicht gefordert, weil zielgerichtete Therapieoptionen noch nicht zur Verfügung standen. Die Relevanz des BRCA1/2-Mutationsnachweises beim HER2/neu negativen frühen Mammakarzinom (HR+/HER2- und tripel-negativen EBC) hat durch die Publikation der OlympiA Studie an Therapiebedeutung gewonnen. Die Ergebnisse der OlympiA-Studie (NCT02032823) haben belegt, dass Patientinnen mit frühem HER2-negativen Mammakarzinom und einer Keimbahnmutation der BRCA1/2-Gene (gBRCAm) von einer einjährigen adjuvanten Therapie mit dem PARP-Inhibitor Olaparib mit einem verbesserten Krankheits- und fernmetastasenfreien Überleben profitieren [4].
Im März 2022 erteilte die FDA die Zulassung von Olaparib für die adjuvante Behandlung von Patientinnen mit einem frühen HER2-negativen Mammakarzinom und einer nachgewiesenen BRCA1/2- Keimbahnmutationen (gBRCA1/2m) nach vorhergehender adjuvanter oder neoadjuvanter Chemotherapie mit Anthrazyklinen und/oder Taxanen sowie einem hohen Rezidivrisiko. Die Europäische Arzneimittelbehörde (EMA) hat im Juni 2022 eine Zulassungserweiterung in Anlehnung an die FDA-Empfehlung beschlossen.
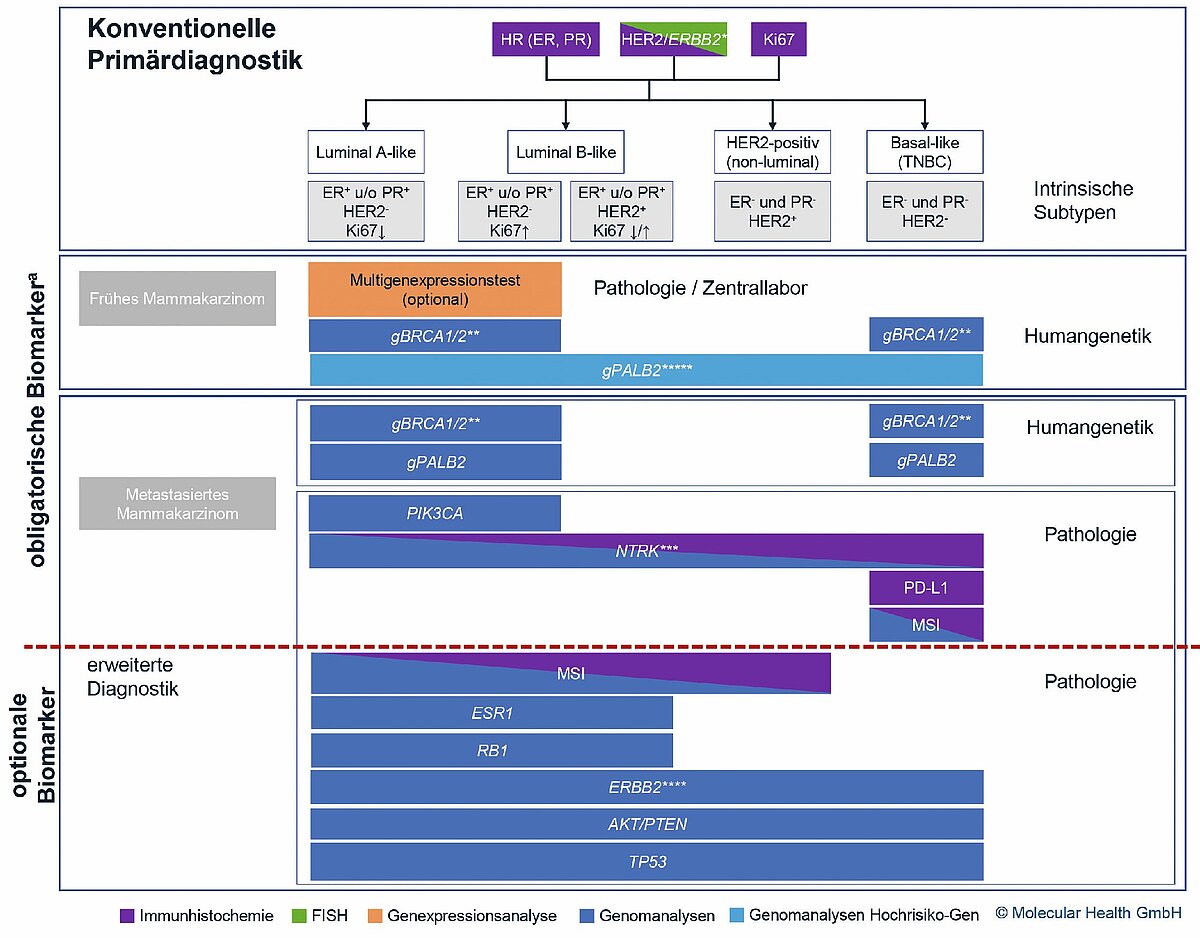
Somit ist die gBRCA1/2m der Biomarker für den Einsatz einer einjährigen oralen Therapie mit Olaparib (2 x 300 mg/die) beim HER2--EBC mit einem erhöhten Rückfallrisiko entsprechend den Einschlusskriterien der OlympiA Studie. In jedem Fall sollte für die bisher nicht betroffenen Familienmitglieder eine humangenetische Beratung und ggf. Testung empfohlen werden. Eine Auswahl therapierelevanter Biomarker beim Mammakarzinom ist in Abb. 1 dargestellt.
Fazit
- Zum Ausschluss eines hereditären Mammakarzinoms sollte die Familienanamnese an Hand des Fragebogens der DKG und der LÄKWL genutzt werden (https://www.onkozert.de/informationen-zertifizierung/hinweise-downloads/checkliste_erbliche_belastung_brust_gyn-210212)
- Multigen-Expressionstests können bei luminalen Karzinomen die Therapieentscheidung für oder gegen eine adjuvante Chemotherapie durch eine quantitative Abschätzung des Rückfallrisikos unterstützen.
- Beim tripel-negativen Mammakarzinom sollte in frühen Stadien für allen Betroffenen < 60. Lebensjahr der BRCA1/2-Mutationsstatus bestimmt werden, um das Vorliegen einer hereditären Erkrankung abzuklären.
- Der Nachweis einer Keimbahnmutation im BRCA1/2-Gen ist der Biomarker für alle HER2/neu negativen Mammakarzinome nach einer neo-adjuvanten Chemotherapie mit einem erhöhten Rückfallrisiko für eine einjährige Therapie mit dem oralen PARP-Inhibitor Olaparib.
Obligatorische molekulare Diagnostik beim metastasierten Mammakarzinom (MBC)
Prädiktive Biomarker für in klinischer Entwicklung befindliche Wirkstoffe
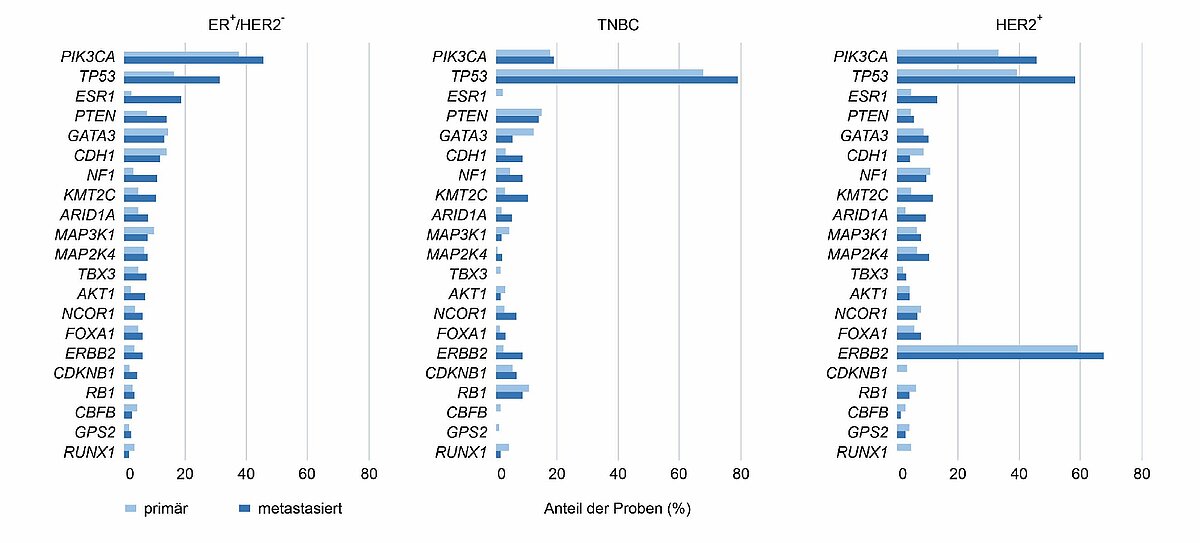
Fehlen Standardtherapieoptionen, können mithilfe umfassender Genomanalysen prädiktive Biomarker für potenziell wirksame Therapieansätze und geeignete klinische Studien identifiziert werden. Bei der vergleichenden Suche nach biologisch relevanten Treibermutationen zeigen sich im Vergleich zwischen dem EBC und dem MBC überraschenderweise in den drei relevanten Gruppen (HR+/HER- versus TNBC versus HER2+) keine großen Abweichungen bei den klinisch relevanten Mutationen, die bei ca. 40 % aller Mammakarzinome nachgewiesen werden können.
Bei fehlenden Standardtherapieoptionen können mithilfe umfassender Genomanalysen prädiktive Biomarker für potenziell wirksame Therapieansätze oder den Einschluss in geeignete klinische Studien identifiziert werden. ERBB2-Mutationen, welche unabhängig von einer Genamplifikation bei 5–6 % der Mammakarzinome nachgewiesen wurden, können auf eine mögliche Wirksamkeit der Tyrosinkinase-Inhibitoren Neratinib bzw. Lapatinib hinweisen. Bei den in 5,8 bzw. 12,3 % der Tumorproben detektierten Genveränderungen in AKT und PTEN können die AKT-Inhibitoren Capivasertib oder Ipatasertib den PIK3CA-Signalweg blockieren [15].
Der Nachweis einer Mikrosatelliteninstabilität (MSI) bei 1,7 % der Mammakarzinome weist auf die potenzielle Wirksamkeit von Immuncheckpoint-Inhibitoren in dieser kleinen Gruppe hin [16]. Für Pembrolizumab besteht eine Pantumor-Zulassung der FDA als „late line therapy“ für solide Tumoren mit MSI. Inaktivierende TP53-Mutationen sind beim metastasierenden Mammakarzinom häufig. Derzeit befinden sich verschiedene Substanzen in klinischer Entwicklung, welche die Funktion des Tumorsuppressorgens direkt oder indirekt wiederherstellen [17].
Die molekulare Diagnostik beim metastasierten Mammakarzinom gehört zur Routine der Therapieplanung. Gemäß den AGO-Empfehlungen 2022 ist die Biopsie einer Metastase immer empfohlen, um einerseits die Dignität der Läsion zweifelsfrei zu klären und ebenso den aktuellen Subtyp zu kennen und ggf. weitere molekulare Marker zu bestimmen. Alterationen in den Genen BRCA1 und BRCA2, PIK3CA sowie optional NTRK1–3 haben bereits heute therapeutische Implikationen [3, 4] und sind als obligate Biomarker im metastasierten Stadium zu verzeichnen (vgl. Abb. 2).
BRCA1/2-Diagnostik
Patientinnen mit HER2/neu-negativem, lokal fortgeschrittenen oder metastasiertem Brustkrebs können bei Vorliegen einer Keimbahnmutation in BRCA1/2 mit PARP-Inhibitoren behandelt werden [5,6]. Bei der Diagnostik einer Keimbahnmutation im BRCA1/2-Gen für eine Therapie mit einem PARP-Inhibitor handelt es sich um diagnostische genetische Untersuchung nach dem Gendiagnostikgesetz (GenDG) [7]. Diese erfolgt an einer Blutprobe und kann von jedem approbierten Arzt veranlasst werden. Im Falle einer Keimbahnmutation sollte eine humangenetische Beratung der Familienangehörigen angeboten werden. Dieses Therapie- und Beratungsangebot gilt für das TNBC und die luminalen Karzinome in der metastasierten Situation gleichlautend, um gBRCA1/2m nachzuweisen und die Option einer PARP-Inhibitor Therapie nicht zu übersehen [2, 3]. Leider wird dieses Angebot in der Routineversorgung noch viel zu selten umgesetzt.
Bestimmung weiterer Risikogene bei positiver Familienanamnese oder gBRCA1/2m beim MBC
Im Rahmen einer humangenetischen Beratung oder bei Nachweis einer gBRCA1/2-Mutation kann eine ergänzende Paneltestung von Risikogenen erforderlich sein. Das Panel enthält Gene mit bekannt hohem Risiko für die Erkrankung an Mammakarzinom (BRCA1, BRCA2, PALB2) sowie Gene mit moderat erhöhtem Risiko (ATM, CHEK2, BARD1, RAD51C, RAD51D) [2, 3, 9]. Nur so können auch nicht BRCA1/2-assoziierte Karzinom-Syndrome ausgeschlossen werden (vgl. Tab. 1).
| Tab. 1: Nicht-BRCA1/2-assoziierte Karzinom-Syndrome | ||
| Syndrom | Gene | Karzinomrisiko |
| Li Fraumeni | TP53 | Brust, Endometrium, Kolorektal, Dünndarm, Magen, Leber, Haut, Osteosarkom, Weichteilsarkom, Urogenital, zentrales Nervensystem (ZNS), adrenokortikales Karzinom (ACC), Lymphom, Lunge |
| Cowden | PTEN | Brust, Endometrium, Schilddrüse, Kolorektal, Niere, Melanom |
| erbliches diffuses Magenkrebssyndrom | CDH1 | erblicher diffuser Magenkrebs, lobulärer invasiver Brustkrebs |
| Peutz-Jeghers | STK11/LKB1 | Kolorektal, Dünndarm, Magen, Pankreas, Hoden, Endometrium |
| Lynch | MLH1, MSH2, MSH6, PMS2, ECAM | Endometrium, Ovar, Kolorektal, Dünndarm, Magen, Leber, Niere, Pankreas, Urogenital, ZNS |
PIK3CA-Mutationsdiagnostik
Beim metastasierten Mammakarzinom findet man in ca. 40 % eine Mutation im PIK3CA-Gen, die somit die häufigste Mutation darstellt [10]. Bei Nachweis einer solchen Mutation besteht seit der Zulassung des PIK3CA-Inhibitors Alpelisib eine sehr gute Therapieoption in Kombination mit dem Östrogenrezeptor-Antagonisten Fulvestrant beim HR+/HER2- endokrin vortherapierten metastasierten Mammakarzinom. Die Mutationsdiagnostik kann einerseits an Tumor- oder Metastasengewebe durchgeführt werden oder an freier Tumor-DNA aus einer Plasmaprobe (Liquid Biopsy). Hierbei ist zu beachten, dass für die Testung an Gewebe möglichst aktuelles Tumor- oder Metastasengewebe verwendet wird. Ein negatives Testergebnis einer Liquid Biopsy sollte bei zu geringer ctDNA-Menge und niedrigen Allelfrequenzen – wo immer möglich – an einer Gewebeprobe überprüft werden.
Bedauerlicherweise muss derzeit die Kostenübernahme für Alpelisib beantragt und der Wirkstoff über die internationale Apotheke angefordert werden. Antragsunterlagen sind bei der AGO Organkommission Mamma erhältlich [11].
Neurotrophe Tyrosin-Rezeptor-Kinase (NTRK)-Gene – NTRK-Genfusionen
Ein molekulargenetischer Marker, der derzeit in den Fokus rückt und eine zielgerichtete, personalisierte Behandlung von Tumoren ermöglicht, betrifft Fusionen der neurotrophen Tyrosin-Rezeptor-Kinase (NTRK)-Gene. Die NTRK-Inhibitoren Larotrectinib und Entrectinib sind indikationsübergreifend bei soliden Tumoren mit NTRK1–3-Fusion und lokal fortgeschrittener oder metastasierter Erkrankung zugelassen. Die Genveränderungen sind selten und diese agnostische Analytik ist aufwendig. Wird aber eine entsprechende Genveränderung nachgewiesen, können die Patientinnen lang anhaltend von der Therapie mit NTRK-Inhibitoren profitieren. Der Nachweis der Fusionen erfolgt meist mithilfe von RNA-basierten Assays, ggf. nach Vorscreening zum Nachweis der Expression der NTRK-Proteine 1, 2 oder 3 mittels Immunhistochemie [12].
Beim Mammakarzinom sind NTRK-Fusionen sehr selten. Aktuelle Untersuchungen deuten darauf hin, dass Genfusionen mit NTRK-Beteiligung bisher ausschließlich beim sekretorischen (juvenilen) Mammakarzinom nachgewiesen werden, welches unter 1 % der invasiven Mammakarzinome ausmacht [13]. Der klinische Verlauf beim sekretorischen Mammakarzinom ist meist indolent und meist mit einer günstigen Prognose assoziiert, obwohl es generell zum Spektrum der tripel-negativen, basal-like Typen gezählt wird. Nur in diesem Fall ist daher die Anforderung einer entsprechenden Analytik sinnvoll. Der molekulargenetische Nachweis der ETV6- NTRK3-Genfusion, hervorgerufen durch eine balancierte Translokation t(12;15), sichert letztlich die Diagnose [14].
Marker für eine primäre oder sekundäre endokrine Resistenz
Ganzgenom-Analysen (Whole Genome Sequencing, WGS) des primären und metastasierten Mammakarzinoms zeigen ein unterschiedliches Mutationsprofil. Aufgrund des durch die Therapie ausgelösten zellulären Selektionsdrucks finden sich beim endokrin behandelten luminalen Subtyp vermehrt Mutationen in ESR1, beim HER2-positiven Subtyp Mutationen in ERBB2, vgl. Abb. 2 [29]. ESR1-Mutationen, die zu einer ligandenunabhängigen Aktivierung des Östrogenrezeptors führen, sind unter Aromatasehemmern, im Gegensatz zu Tamoxifen, häufig beobachtete Resistenzmechanismen gegen endokrine Therapien [18]. Bei bestehender Indikation zur Fortführung einer endokrinen Therapie sollte der selektive Östrogen-Degrader (SERD) Fulvestrant bei Vorliegen einer ESR1-Mutation eingesetzt werden. Eine Abklärung durch Genomsequenzierung unter Abdeckung relevanter Genbereiche kann die Wahl der Folgetherapie unterstützen. Auch eine primäre Resistenz gegen CDK4/6-Inhibitoren z. B. aufgrund eines Verlusts des RB1-Gens oder des Vorliegens einer BRCA2-Mutation könnte so erkannt werden [19].
Spätestens bei Resistenz gegen CDK4/6- Inhibitoren ist eine umfassende genomische Analytik sinnvoll, um Resistenzmechanismen abzuklären, und die Therapieausrichtung zu adjustieren. In der PADA-1-Studie konnte erstmals prospektiv gezeigt werden, dass unter einer laufenden endokrinen Therapie mit einem Aromatasehemmer der Nachweis einer ESR1 Mutation in der Liquid Biopsie bei einem frühzeitigen Therapiewechsel auf einen SERD + CDK4/6-Inhibitor das krankheitsfreie Überleben signifikant verbessern kann, bevor klinische Symptome der Progression erkennbar waren [20].
Wachsende Herausforderungen an die Interpretation NGS-basierter Analysen
Die Interpretation von Next-Generation-Sequencing-(NGS-)Daten ist hoch komplex und erfordert eine hohe Expertise von zahlreichen Experten. Durch die parallele Sequenzierung von Millionen kurzer DNA-Fragmente generiert NGS eine enorme Menge an Rohdaten, die analysiert werden müssen, um das Vorliegen einer Mutation zu bestätigen oder auszuschließen. Die gewonnenen Sequenzdaten werden bioinformatisch mit einem humanen Referenzgenom abgeglichen, um abweichende Veränderungen zu erfassen. Die Verlässlichkeit der Informationen wird unter Einbeziehung wichtiger Qualitätsparameter wie z. B. Variantenallelfrequenz, Coverage und Tumorzellgehalt von der Molekularpathologie bzw. Humangenetik bewertet. Anschließend wird die therapeutische Bedeutung der detektierten Genveränderungen in der Regel anhand von öffentlich zugänglichen Datenbanken recherchiert (z. B. ClinVar) und die Varianten nach ihrer Bedeutung in fünf Stufen klassifiziert. Für die Therapie beispielsweise mit PARP-Inhibitoren müssen BRCA1/2-Varianten der Klasse 4 (wahrscheinlich pathogen) oder 5 (sicher pathogen) nachgewiesen werden. Grundsätzlich werden sämtliche Genvarianten in fünf Klassen unterteilt:
- Klasse 1: nicht pathogen
- Klasse 2: wahrscheinlich nicht pathogen
- Klasse 3: Variante unklarer Signifikanz (VUS)
- Klasse 4: wahrscheinlich pathogen
- Klasse 5: pathogen
Sind die gefundenen Varianten klassifiziert, muss eine Interpretation der klinischen Bedeutung der Varianten erfolgen. Dazu sollten internationale Klassifizierungssysteme wie der AMP/CAP- oder ESCAT-Score verwendet werden [21, 22]. Mit wachsender Komplexität der NGS-Analysen werden vermehrt seltene und/oder neue Genveränderungen detektiert. Die Klassifizierung dieser Varianten bindet zunehmend Zeit- und Personalressourcen. Aufwendige Recherchen zur Abklärung möglicher diskrepanter Ergebnisse und der Zuordnung der klinischen Relevanz können erforderlich sein. Hier werden intelligente Softwarelösungen wie z. B. Molecular Health Guide, Pierian DX oder QCI Interpret verwendet, die die zeitraubenden Recherchen in biomedizinischen Datenbanken für die Variantenklassifikation (sog. Annotation) automatisiert durchführen und außerdem die klinische Interpretation unterstützen und standardisieren, vgl. Abb. 3 (nur online) [23–25].
Perspektiven der molekularen Diagnostik in der Krankenversorgung
Die Anforderungen an molekulare Analysen und die klinische Relevanz detektierter Varianten müssen klaren Evidenzen zugeordnet werden, wie eine aktuelle Auswertung der Studie SAFIR02 (NCT02299999) beim endokrin resistenten Mammakarzinom zeigt. Nur Patientinnen mit genomischen Aberrationen von hoher therapeutischer Relevanz (sog. ESCAT Level I/II) profitierten von einer entsprechend gewählten zielgerichteten Therapie im Vergleich zur Chemotherapie [26].
Mit zunehmendem Bedarf an ausgedehnten molekularen Analysen wird die Bedeutung der molekularen Tumorboards für das Mammakarzinom zunehmen. Dies stellt Anforderungen an die rechtzeitige Schaffung der entsprechenden Infrastruktur. An Standorten, an denen eine entsprechende Struktur noch nicht flächendeckend angeboten werden kann, ist daher die Bildung von Netzwerken mit Zugang zu molekularen Tumorboards sinnvoll. Im Rahmen der klinischen Netzwerkstruktur des OnKoNetRhein Main (www.onkonetrheinmain.de) sind diese infrastrukturellen Voraussetzungen seit Jahren etabliert und bieten einen interdisziplinären und sektorenübergreifenden Zugriff auf molekulare Tumorboards und verschiedene Tumorboards in der Region.
Prof. Dr. med. Christian Jackisch, Chefarzt der Klinik für Gynäkologie und Geburtshilfe, Sana Klinikum Offenbach, E-Mail: christian.jackisch@sana.de
Prof. Dr. med. Peter J. Wild, Direktor des Dr. Senckenbergischen Instituts für Pathologie am Universitätsklinikum Frankfurt sowie Universitätsklinikum Frankfurt MVZ GmbH –Wildlab
Die beiden Autoren sind Mitglieder des OnkoNet Rhein Main e. V., im Internet: www.onconetrheinmain.de
Multiple Choice-Fragen
Die Multiple Choice-Fragen zu „Molekulare Tumordiagnostik beim Mammakarzinom“ von Prof. Dr. med. Christian Jackisch und Prof. Dr. med. Peter J. Wild finden Sie auch im Mitglieder-Portal (https://portal.laekh.de) sowie am Ende dieser Seite unter „PDF herunterladen“. Die Teilnahme zur Erlangung von Fortbildungspunkten ist ausschließlich online über das Mitglieder-Portal vom 25. Dezember 2022 bis 24. Juni 2023 möglich. Die Fortbildung ist mit zwei Punkten zertifiziert. Mit Absenden des Fragebogens bestätigen Sie, dass Sie dieses CME-Modul nicht bereits an anderer Stelle absolviert haben. Dieser Artikel hat ein Peer-Review-Verfahren durchlaufen. Nach Angaben der Autoren sind die Inhalte produkt- und/oder dienstleistungsneutral, etwaige Interessenkonflikte werden im Literaturverzeichnis (online) offengelegt.
Abkürzungsverzeichnis | |
AGO | Arbeitsgemeinschaft Gynäkologische Onkologie |
AMP-Score | Amputee Mobility Predictor |
BRCA1/BRCA2 | Breast-cancer-1 (BRCA1)- oder BRCA2-Gen |
ctDNA | Circulating tumor DNA, DNA-Fragmente von malignen Tumoren, die sich im peripheren Blut nachweisen lassen |
CDK4/6- Inhibitoren | Inhibitoren der Cyclin-abhängigen Kinasen |
DKG | Deutsche Krebsgesellschaft |
DNA | Desoxyribonukleinsäure |
EBC | early breast cancer, frühes Mammakarzinom |
EMA | Europäische Arzneimittelbehörde |
ER | Östrogenrezeptor |
ERBB2 | Genname von HER2 |
ESR1-Mutation | Mutationen im Östrogenrezeptor |
FDA | Food and Drug Administration, US-amerikanische Arzneimittelbehörde |
GenDG | Gendiagnostikgesetz |
HER2 | Human Epidermal Growth Factor Receptor 2 |
HR | Hormonrezeptor |
HR+/HER2- | luminale Karzinome |
KI | künstliche Intelligenz |
NTRK | Neurotrophe-Tyrosin-Rezeptor-Kinase-Gen |
PARPi | Poly (ADP-Ribose) Polymerase (PARP)-Inhibitoren |
PR | Progesteronrezeptor |
TNBC | triple negativer Brustkrebs |
Die Literaturhinweise finden Sie unter "PDF-Download" in der PDF-Version dieses Artikels.
Erklärung zu Interessenkonflikten
Prof. Dr. Christian Jackisch hat Beratungsgebühren und Honorare für Vorträge von AstraZeneca, Pfizer, Celgene, Exact Sciences, Lilly, Pierre Fabre, Roche SanofiGenzyme, Molecular Health, Gilead, medupdate, StreamedUp erhalten. Die Forschung wurde von ExactScienes und Roche unterstützt. Mitgliedschaften: Deutsche Gesellschaft für Gynäkologie und Geburtshilfe, Arbeitsgemeinschaft für Gynäkologische Onkologie e. V., Deutsche Krebsgesellschaft e. V., Hessische Krebsgesellschaft e. V.
Prof. Dr. Peter J. Wild hat Beratungsgebühren und Honorare für Vorträge von Bayer, Janssen-Cilag, Novartis, Roche, MSD, Astellas Pharma, Bristol-Myers Squibb, Thermo Fisher Scientific, Molecular Health, Guardant Health, Sophia Genetics, Qiagen, Eli Lilly, Myriad, Hedera Dx und Astra Zeneca erhalten. Die Forschung wurde von Astra Zeneca und Roche unterstützt. Mitgliedschaften: Deutsch Gesellschaft für Pathologie, Europäische Gesellschaft für Pathologie, Internationale Akademie für Pathologie, Bundesverband Deutscher Pathologen.